
By Zannanza's history channel | 2020-09-30 | 自然科學

DNA測序技術,是遺傳學家分析人類及其他生物基因的重要工具。自從1950年代DNA雙螺旋結構被Watson and Crick發現以來,基因組研究已經走了很遠的路。由2003年人類基因圖譜計劃完成,直到最近人類基因組測序的費用跌破$1000美金,並開始應用響醫療領域,DNA測序技術的進步已經遠遠超越很多人的預期。這裡,會簡介一下到底DNA是甚麼,和DNA測序及相關技術的原理。
DNA:絕大部分生物所使用的遺傳物質
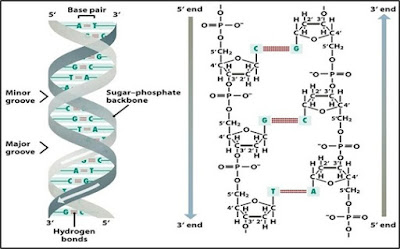
DNA,中文名稱脫氧核醣核酸,是絕大部分生物所使用的遺傳物質。除了少量病毒使用RNA作為遺傳物質之外,其他高等生物均無一例外使用DNA作遺傳用途。DNA是長長的鏈狀結構,由兩條反平行(anti-parallel,即是平行但方向調轉,其中一條5'->3',另一條方向則為3'->5')的長鏈狀分子所組成,兩條鏈互相纏繞而成雙螺旋結構(double helix)。每條DNA由醣-磷酸骨架(Sugar-phosphate backbone)同鹼基(base)組成,醣-磷酸骨架連接每個鹼基形成長鏈,而鹼基則負責儲存遺傳信息。鹼基基本上有分四種:A、T、C、G。鹼基有一種特性叫鹼基對(base pairing),即是一條鏈上的A相對反方向鏈的相同位置就是T、C則對G,調返轉亦都一樣。而DNA鏈上ATCG四種鹼基的排序,則儲存著生物遺傳相關的信息。相比起化學上較不穩定的RNA,DNA是化學上穩定的分子,不容易降解和被破壞,這個亦都是大部分生物將用作遺傳用途的原因,因為可以將遺傳信息穩定地由一代傳到下一代。
在真核生物的細胞中,DNA遺傳物質主要集中響細胞核(nucleus)入面,而DNA在細胞核入面不是以裸分子(naked DNA)形態存在,而是同組織蛋白(histone)和其他蛋白質結合組成染色體(chromosome)。在細胞分裂期間,染色體以聚結形態出現,而在非分裂期間,則以染色質(chromatin)形態出現。基因組入面,DNA鹼基對的數量按不同生物品種有所區別,以人類基因組為例,則有23對染色體,大約30億對鹼基對。
染色質在細胞核內的結構
在細胞分裂期間染色質凝聚成為染色體
大約認識了DNA的化學結構和特點,下集正式開始介紹如何為DNA鹼基測試排序
DNA測序的鼻祖:鏈終止法,或稱Sanger法

鏈終止法,或稱Sanger法,於1970年代由弗雷德里克·桑格(Frederick Sanger)及其同事所創,至今仍廣為應用的DNA測序法。Sanger法首先必須按照DNA已知的序列設計一個譯引子(primer),在聚合酵素連鎖反應法(Polymerase Chain Reaction,PCR)入面,DNA聚合酵素會模仿自然界中DNA複製(DNA replication),從譯引子既3'羥基 -OH group開始,利用反應物當中所含的核苷酸(nucleotide,即DNA長鏈的組成部分),延長一條新的反方向DNA長鏈,最終完成一條新的雙螺旋。鏈終止法顧名思義,就是利用延長鏈過程當中中止反方向長鏈的生成,產生出一堆不同長度的DNA鏈。然後再利用不同長度DNA鏈可被分離的特性,從而探測出DNA終止位置的每個鹼基到底是A、T、C還是G。這個就是超簡化的Sanger法基礎原理。
到底Sanger法實際操作是怎樣進行呢?
首先,試管入面要有準備用作測序的DNA(template DNA),譯引子(primer)。並且提供延長DNA鏈所需用的building block,即四種核苷酸dATP、dTTP、dCTP、dGTP(統稱dNTP),以及延長反方向DNA鏈的催化劑(catalyst),即DNA聚合酵素(DNA polymerase)。另外,試管入面仲會加入一種特別的核苷酸,這個核苷酸缺乏位於核醣(ribose)第三個炭原子的羥基(3' hydroxyl group,或稱3' -OH group),因此被稱為雙脫氧核苷酸(dideoxynucleotide)。雙脫氧核苷酸係PCR反應當中終止反方向DNA鏈延長所用的抑制劑,負責停止延長反應。由於它缺乏3' -OH group,DNA聚合酵素當加入一個雙脫氧核苷酸後無法繼續像正常一樣將另一個核苷酸的磷酸根跟3' -OH group反應,因此該DNA鏈不再延長。雙脫氧核苷酸總共有四種,ddATP、ddTTP、ddCTP、ddGTP(統稱ddNTP)。而雙脫氧核苷酸上則以放射性原子(radioactive atom)或染色劑(dye)作標記,以便不同長度的DNA鏈按長度分離後偵測出該終止位置的鹼基類別。
雙脫氧核苷酸結構和一般核苷酸結構,雙脫氧核苷酸缺乏3' -OH group,因此當DNA鏈加入雙脫氧核苷酸後,則無法再繼續延長
Sanger法的工作流程
有齊所有反應物之後,試管會被放置到PCR機器當中,PCR機器首先會利用高溫將DNA雙螺旋當中的兩條長鏈分開(denaturation),然後會降溫以便譯引子(primer)及聚合粘著準備要測序的DNA長鏈。然後再度升溫,讓聚合酵素開始工作,將dNTP加入正在生成中的長鏈上。在過程中,聚合會隨機加入ddNTP從而令DNA延長過程中止。PCR機器會不斷重覆升溫、降溫的循環,當若干循環後,一大堆不同長度的DNA鏈就會出現在試管內。然後試管內容物會被移送到DNA測序儀內。DNA測序儀內有一個裝有緩衝溶液的水缸,一塊滿佈小孔的凝膠,及位於水缸兩端的陰陽兩極。由於DNA分子當中擁有帶負電荷的磷酸根(phosphate group),當通電後DNA會在凝膠內向正極移動。越長的DNA越重,移動得越慢,而較短的DNA則較輕,移動得越快,因此通電後長短不一的DNA就會按長度分開。在過程中,DNA測序儀會探測經過的ddNTP上的螢光染劑。由於ddATP、ddTTP、ddCTP、ddGTP四種ddNTP上所用不同顏色的螢光染劑,機器便可以區分開經過的DNA鏈到底終止位置的鹼基到底是A、T、C或是G。利用Sanger法,我們可以得到800-1000個鹼基的排列資訊,我們稱之為「讀長」(read length)。但等一等,人類基因組有成30億對鹼基對,如果Sanger法只能產生800-1000個鹼基的讀長,那怎麼可以用來測序整個人類完整基因組既鹼基排列呢?
答案:人類基因圖譜是利用很多很多短的、800-1000個鹼基長度的DNA斷片(fragments)拼接而成。因此學者必須將人類基因組的超長DNA鏈進行降解、切割成可測序的長度,並對大量的短DNA斷片進行測序,最後利用不同斷片之間的重疊序列,好像砌puzzle一樣拼合而成一個完整的基因組。測序一個人類基因組,如果利用Sanger法大約需要6倍的覆蓋(6 fold coverage),即是要測序整個基因組長度既DNA 6次,先至可以產生足夠重疊序列用作拼合成一個完整的基因組。
人類基因圖譜係利用重疊序列拼合成一個完整的基因組
Sanger法是1990年代人類基因圖譜計劃所用的測序技術,但Sanger法無法大規模平行操作,因此單鹼基測序成本非常高昂。在人類基因圖譜計劃當中,冷泉港實驗室大量的老式3730型測序儀日以繼夜地運作,足足運作了十三年、耗資30億美金先至將第一個人類基因組序列完成。到了2000年代中葉,新測序技術的發明先至將DNA測序既價格開始降低。新測序技術(Next generation sequencing technology),或稱次世代測序技術的誕生,最終形成了一場革命,徹底改變了DNA測序的成本、市場同應用。關於這次世代測序技術,就容下次再說。
大量的老式3730型測序儀日以繼夜地運作先至將第一個人類基因組序列完成
本文經授權轉載自<Zannanza's history channel>專頁

筆者是膠登及連登會員,《聚言時報》、《故事》、《Fortune Insight》專欄作家。文、理、商科背景俱備,但自覺「周身刀,冇張利」。筆者筆名來自公元前14世紀期間,被西臺帝國國王蘇庇路里烏瑪一世派往埃及迎娶埃及王后安克姍海娜曼卻神秘消失在兩國邊界的西臺王子塞那沙。筆者專寫古典世史,希臘、羅馬、古代近東、氣候、科技、建築及經濟史均有涉獵,著作包括《羅馬是怎樣建成的》、《漫談荷馬史詩中的歷史背景》、《西臺帝國興衰史》、《亞歷山大與希臘化時代》等。